Cet article a été co-écrit avec Stephen Aylward, Matt McCormick, Brianna Major de Kitware et Justin Kirby du Frederick National Cancer Research Laboratory (FNLCR).
Amazon SageMaker Studio Lab offre un accès gratuit à l’environnement de développement de machine learning (ML) à toute personne disposant d’une adresse e-mail. À l’instar d’Amazon SageMaker Studio, Studio Lab vous permet de personnaliser votre environnement Conda et de créer des ordinateurs portables JupyterLab v3 pour un processeur et un processeur graphique évolutifs, avec un accès facile aux derniers outils de productivité de la science des données et aux bibliothèques open source. En plus de cela, les comptes Studio Lab gratuits incluent un minimum de 15 Go de stockage permanent, vous permettant de conserver vos projets et vos dépenses sur plusieurs sessions et vous permettant instantanément de reprendre là où vous vous étiez arrêté et même de partager votre travail et votre travail en cours. environnements avec les autres.
L’un des enjeux majeurs auquel est confrontée la communauté de l’imagerie médicale est de savoir comment permettre aux chercheurs d’expérimenter et d’explorer ces outils essentiels. Pour résoudre ce défi, les équipes AWS ont travaillé avec Kitware et le Frederick National Cancer Research Laboratory (FNLCR) pour rassembler trois ressources d’IA clés pour l’imagerie médicale dans Studio Lab et la communauté entièrement open source JupyterLab :
Ces outils et données se combinent pour permettre aux chercheurs en imagerie médicale IA de développer rapidement et d’évaluer de manière exhaustive des algorithmes d’apprentissage en profondeur cliniquement prêts dans un environnement complet et facile à utiliser. Les membres de l’équipe du FNLCR et de Kitware ont collaboré pour créer une série de blocs-notes Jupyter qui illustrent les flux de travail courants pour accéder et visualiser par programme les données TCIA. Ces blocs-notes utilisent Studio Lab pour permettre aux chercheurs d’exécuter des blocs-notes sans avoir à configurer leur propre environnement de développement Jupyter local. Vous pouvez rapidement explorer de nouvelles idées ou intégrer votre travail dans des présentations, des ateliers et des didacticiels lors de conférences.
L’exemple suivant montre Studio Lab exécutant un bloc-notes Jupyter qui télécharge les données d’IRM de la prostate à partir de TCIA, les segmente à l’aide de MONAI et affiche les résultats à l’aide d’itkWidgets.

Bien que vous puissiez facilement exécuter des expériences et des démonstrations plus petites à l’aide des exemples de blocs-notes proposés gratuitement dans cet article de Studio Lab, Amazon SageMaker Studio est recommandé lors de la formation de vos modèles d’images médicales à grande échelle. Amazon SageMaker Studio est un environnement de développement intégré (IDE) basé sur le Web avec des fonctionnalités de sécurité, de gouvernance et de surveillance de niveau entreprise dans lequel vous pouvez accéder à des outils spécialement conçus pour effectuer toutes les étapes du développement de l’apprentissage automatique. Les bibliothèques open source telles que MONAI Core et itkWidgets fonctionnent également sur Amazon SageMaker Studio.
Installer la solution
Pour lire les ordinateurs portables TCIA sur Studio Lab, vous devez créer un compte avec votre adresse e-mail sur le site Web de Studio Lab. Les demandes de compte peuvent prendre 1 à 3 jours pour être approuvées.
Ensuite, vous pouvez suivre les étapes d’installation pour commencer :
- Connectez-vous à Studio Lab et démarrez l’exécution de votre processeur.
- Dans un onglet séparé, accédez au référentiel GitHub des blocs-notes TCIA et choisissez un bloc-notes dans le dossier racine du référentiel.
- Il choisit Ouvrir le laboratoire du studio Pour ouvrir le bloc-notes dans Studio Lab.
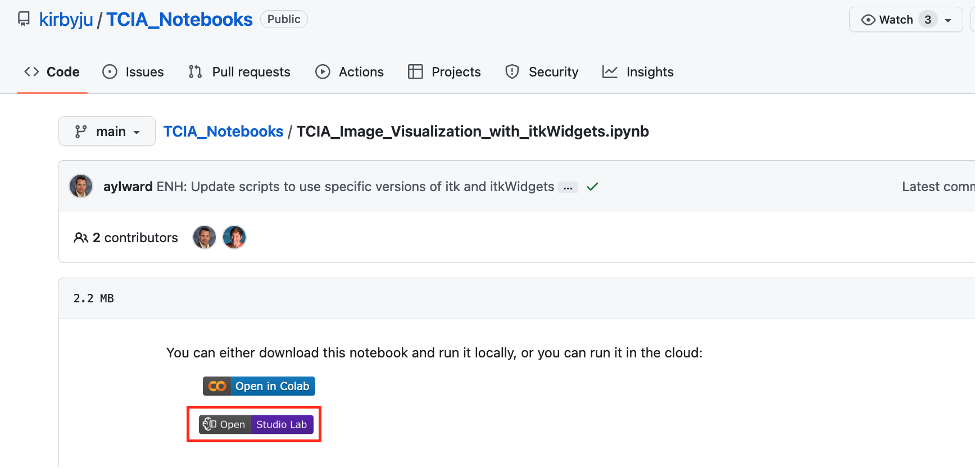
- De retour dans Studio Lab, choisissez Copier dans le projet.

- Dans la nouvelle fenêtre contextuelle JupyterLab qui s’ouvre, choisissez Cloner l’intégralité du dépôt.

- Dans la fenêtre suivante, conservez les valeurs par défaut et choisissez clonage.

- Il choisit Oui Lorsque vous êtes invité à confirmer la construction du nouvel environnement Conda (
medical-image-ai). Il faudra jusqu’à 5 minutes pour créer l’environnement Conda.
Il faudra jusqu’à 5 minutes pour créer l’environnement Conda. - Dans le terminal ouvert à l’étape précédente, exécutez la commande suivante pour installer NodeJS dans un fichier
studiolabEnvironnement Conda, qui est requis pour installer l’extension ImJoy JupyterLab 3 par la suite :conda install -y -c conda-forge nodejs
Nous installons maintenant l’extension ImJoy Jupyter à l’aide de Studio Lab Extension Manager pour permettre des visualisations interactives. L’extension Imjoy permet à itkWidgets et à d’autres processus gourmands en données de communiquer avec les environnements Jupyter locaux et distants, y compris les blocs-notes Jupyter, JupyterLab, Studio Lab, etc. - Dans Extension Manager, recherchez “imjoy” et choisissez installer.

- Assurez-vous de reconstruire le noyau lorsque vous y êtes invité.
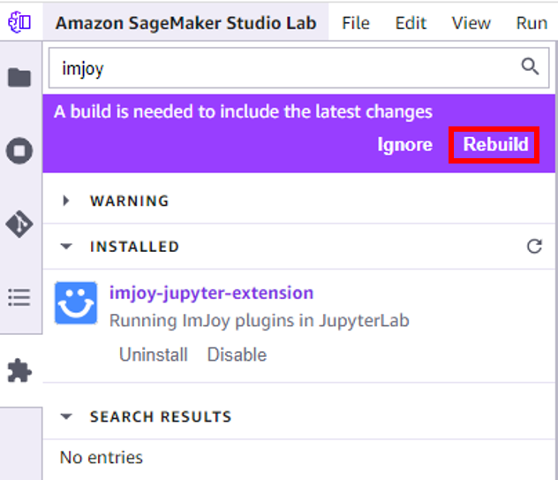
- Il choisit Enregistrer et recharger Lorsque la construction est terminée.

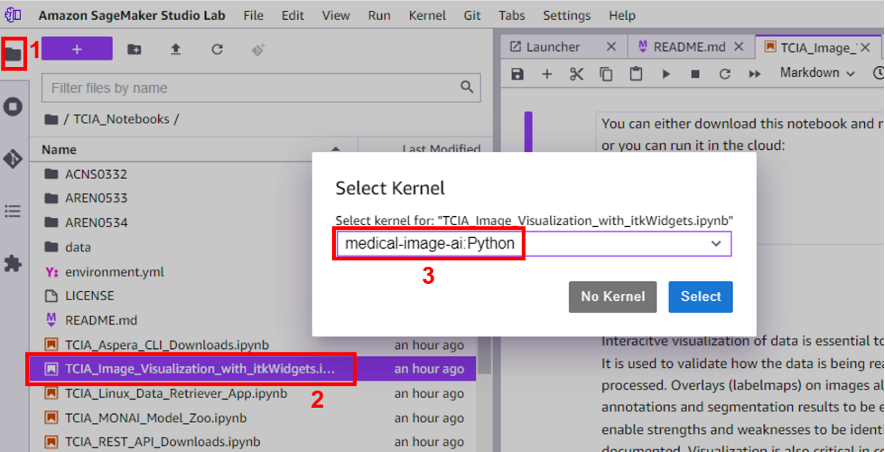
Après avoir installé l’extension ImJoy, vous pourrez voir l’icône ImJoy dans le menu supérieur de vos ordinateurs portables.
Pour le vérifier, accédez à votre navigateur de fichiers et choisissez un fichier TCIA_Image_Visualalization_with_itkWidgets bloc-notes et choisissez medical-image-ai noyau pour l’exécuter.
L’icône ImJoy sera visible dans le coin supérieur gauche du menu du bloc-notes.
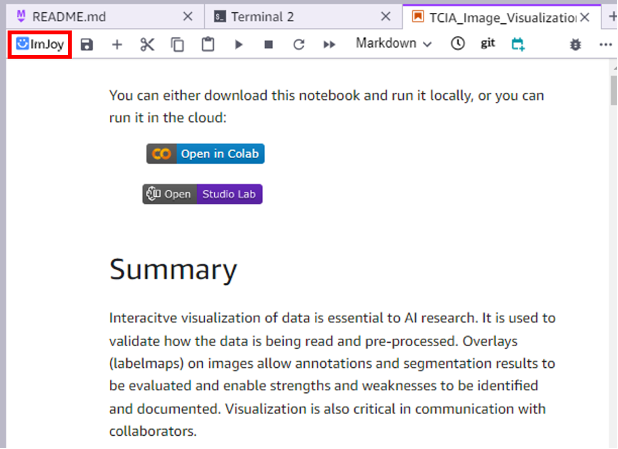
Avec ces étapes d’installation, vous avez installé le medical-image-ai Le noyau Python et l’extension ImJoy comme condition préalable à l’exécution des notebooks TCIA avec itkWidgets dans Studio Lab.
Testez la solution
Nous avons créé un ensemble de blocs-notes et un didacticiel qui montre l’intégration de ces technologies d’IA dans Studio Lab. Assurez-vous de choisir un fichier medical-image-ai Noyaux Python lors de l’exécution de notebooks TCIA dans Studio Lab.
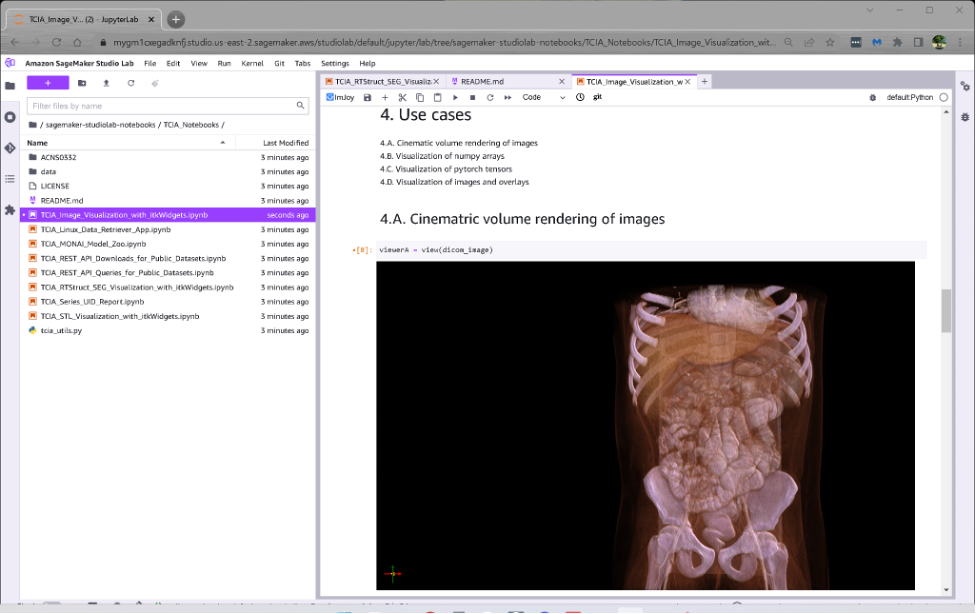
Le premier bloc-notes de SageMaker montre comment télécharger des images DICOM à partir de TCIA et visualiser ces images à l’aide des capacités de rendu au format cinéma d’itkWidgets.
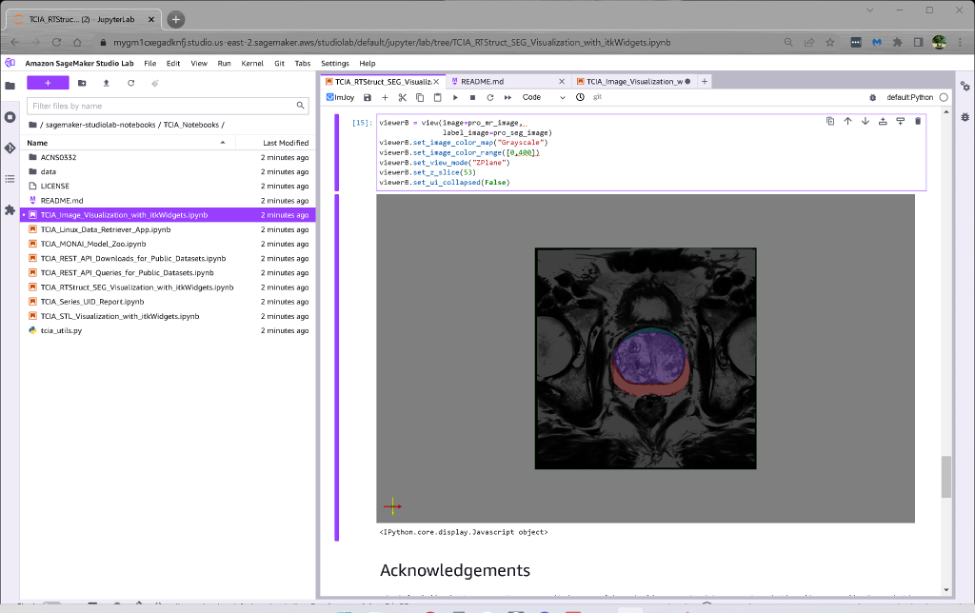
Le deuxième cahier montre comment les annotations d’experts disponibles pour des centaines d’études sur TCIA peuvent être téléchargées en tant qu’objets DICOM SEG et RTSTRUCT, visualisées en 3D ou en superposition sur des tranches 2D, et utilisées pour former et évaluer des systèmes d’apprentissage en profondeur.
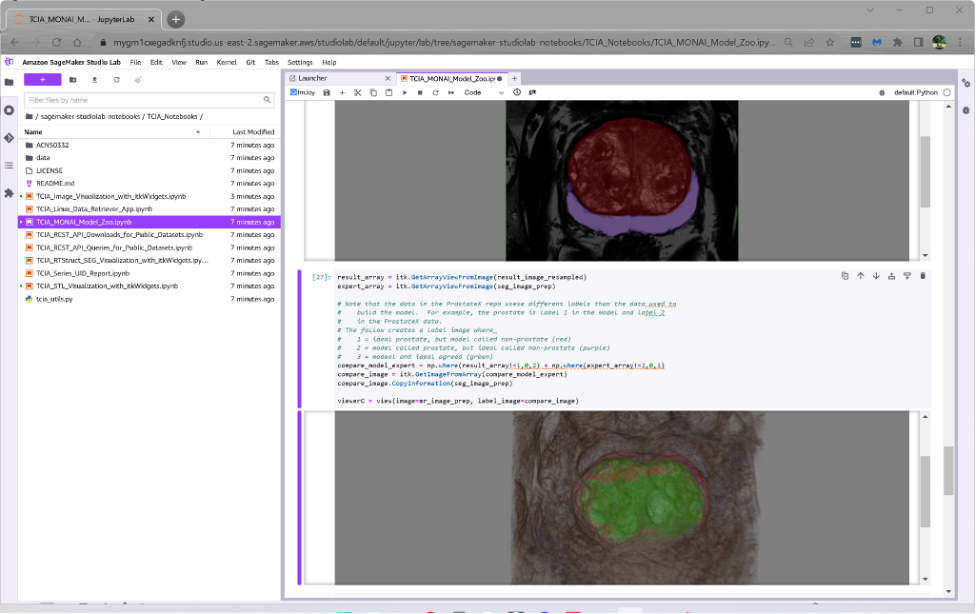
Le troisième cahier montre comment les modèles d’apprentissage en profondeur MONAI disponibles peuvent être téléchargés sur le modèle MONAI ZOO et utilisés pour segmenter (ou les vôtres) les volumes TCIA pour les IRM de la prostate DICOM.
Il choisit Ouvrir le laboratoire du studio sur ces notebooks et sur d’autres notebooks JupyterLab pour les exécuter dans l’environnement Studio Lab disponible gratuitement.
nettoyage
Après avoir suivi les étapes d’installation indiquées dans cet article et créé un medical-image-ai environnement Conda, vous pouvez le supprimer pour économiser de l’espace de stockage. Pour ce faire, utilisez la commande suivante :
conda remove --name medical-image-ai --allVous pouvez également désinstaller l’extension ImJoy via Extension Manager. Sachez que vous devrez reconstruire l’environnement Conda et réinstaller l’extension ImJoy si vous souhaitez continuer à travailler avec les ordinateurs portables TCIA dans votre compte Studio Lab ultérieurement.
Fermez votre onglet et n’oubliez pas de vérifier Arrêter le temps de course Sur la page du projet Studio Lab.
Conclusion
SageMaker Studio Lab est disponible gratuitement pour les communautés de recherche sur l’IA en imagerie médicale et peut être utilisé pour la modélisation d’images médicales IA et la visualisation d’images médicales interactives en association avec MONAI et itkWidgets. Vous pouvez utiliser les données ouvertes TCIA et des exemples de blocs-notes avec Studio Lab lors d’événements de formation, tels que des hackathons et des ateliers. Avec cette solution, les scientifiques et les chercheurs peuvent rapidement mener des expériences, collaborer et innover avec l’IA pour les images médicales. Si vous avez un compte AWS et configurez un domaine SageMaker Studio, vous pouvez également exécuter ces blocs-notes sur Studio en utilisant le noyau Python de science des données par défaut (avec ImJoy-jupyter-extension installé) tout en sélectionnant parmi une variété de types d’instances de calcul.
Studio Lab a également lancé une nouvelle fonctionnalité sur AWS re:Invent 2022 pour prendre des blocs-notes développés dans Studio Lab et les exécuter en tant que tâches par lots selon un calendrier récurrent dans vos comptes AWS. Par conséquent, vous pouvez faire évoluer vos expériences ML au-delà des limites du calcul gratuit de Studio Lab et utiliser des instances de calcul plus puissantes avec des ensembles de données beaucoup plus volumineux sur vos comptes AWS.
Si vous souhaitez en savoir plus sur la façon dont AWS peut aider votre organisation de soins de santé ou de sciences de la vie, veuillez contacter votre représentant AWS. Pour plus d’informations sur MONAI et itkWidgets, veuillez contacter Kitware. De nouvelles données sont ajoutées à TCIA sur une base continue, et nous apprécions vos suggestions et contributions en visitant le site Web de TCIA.
Lectures complémentaires
À propos des auteurs
 Stephen Aylward Il est directeur principal des initiatives stratégiques chez Kitware, professeur adjoint d’informatique à l’Université de Caroline du Nord à Chapel Hill et membre de la MICCAI Society. Le Dr Aylward a fondé le bureau de Kitware en Caroline du Nord, a lancé plusieurs initiatives open source et est maintenant président du conseil consultatif de MONAI.
Stephen Aylward Il est directeur principal des initiatives stratégiques chez Kitware, professeur adjoint d’informatique à l’Université de Caroline du Nord à Chapel Hill et membre de la MICCAI Society. Le Dr Aylward a fondé le bureau de Kitware en Caroline du Nord, a lancé plusieurs initiatives open source et est maintenant président du conseil consultatif de MONAI.
 Matt McCormickIl est ingénieur émérite chez Kitware, où il dirige le développement d’Insight Toolkit (ITK), une boîte à outils scientifique pour l’analyse d’images. Il a été chercheur principal et co-chercheur sur de nombreuses subventions de recherche des National Institutes of Health (NIH), a dirigé des engagements avec les laboratoires nationaux des États-Unis et a dirigé de nombreuses entreprises commerciales fournissant des logiciels avancés pour les dispositifs médicaux. Le Dr McCormick est un ardent défenseur des logiciels open source dirigés par la communauté, de la science ouverte et de la recherche reproductible.
Matt McCormickIl est ingénieur émérite chez Kitware, où il dirige le développement d’Insight Toolkit (ITK), une boîte à outils scientifique pour l’analyse d’images. Il a été chercheur principal et co-chercheur sur de nombreuses subventions de recherche des National Institutes of Health (NIH), a dirigé des engagements avec les laboratoires nationaux des États-Unis et a dirigé de nombreuses entreprises commerciales fournissant des logiciels avancés pour les dispositifs médicaux. Le Dr McCormick est un ardent défenseur des logiciels open source dirigés par la communauté, de la science ouverte et de la recherche reproductible.
 Brianna Major Ingénieur R&D chez Kitware avec une passion pour le développement de logiciels et d’outils open source qui profiteront aux communautés médicales et scientifiques.
Brianna Major Ingénieur R&D chez Kitware avec une passion pour le développement de logiciels et d’outils open source qui profiteront aux communautés médicales et scientifiques.
 jAustin Kirby Il est chef de projet technique au Laboratoire National de Recherche en Cancérologie Frederick (FNLCR). Son travail se concentre sur les moyens de permettre le partage de données tout en préservant la confidentialité des patients afin d’améliorer la reproductibilité et la transparence dans la recherche sur l’imagerie du cancer. Son équipe a fondé The Cancer Imaging Archive (TCIA) en 2010, qui a profité à la communauté de la recherche pour publier plus de 200 ensembles de données liés à des manuscrits, des subventions, des concours et des initiatives de recherche majeures de l’Institut national du cancer. Ces ensembles de données ont été discutés dans plus de 1 500 publications évaluées par des pairs.
jAustin Kirby Il est chef de projet technique au Laboratoire National de Recherche en Cancérologie Frederick (FNLCR). Son travail se concentre sur les moyens de permettre le partage de données tout en préservant la confidentialité des patients afin d’améliorer la reproductibilité et la transparence dans la recherche sur l’imagerie du cancer. Son équipe a fondé The Cancer Imaging Archive (TCIA) en 2010, qui a profité à la communauté de la recherche pour publier plus de 200 ensembles de données liés à des manuscrits, des subventions, des concours et des initiatives de recherche majeures de l’Institut national du cancer. Ces ensembles de données ont été discutés dans plus de 1 500 publications évaluées par des pairs.
 gang fu Il est l’architecte des solutions de soins de santé chez AWS. Il est titulaire d’un doctorat en sciences pharmaceutiques de l’Université du Mississippi et possède plus de dix ans d’expérience dans la technologie et la recherche biomédicales. Il est passionné par la technologie et l’impact qu’elle peut avoir sur les soins de santé.
gang fu Il est l’architecte des solutions de soins de santé chez AWS. Il est titulaire d’un doctorat en sciences pharmaceutiques de l’Université du Mississippi et possède plus de dix ans d’expérience dans la technologie et la recherche biomédicales. Il est passionné par la technologie et l’impact qu’elle peut avoir sur les soins de santé.
 Alex Lime Il est responsable du développement commercial pour l’imagerie médicale chez AWS. Alex identifie et met en œuvre des stratégies de mise sur le marché avec des partenaires d’imagerie et dirige le développement de solutions pour accélérer la recherche en imagerie médicale basée sur l’IA/ML dans le cloud. Il est passionné par l’intégration des frameworks ML open source avec la pile AWS AI/ML.
Alex Lime Il est responsable du développement commercial pour l’imagerie médicale chez AWS. Alex identifie et met en œuvre des stratégies de mise sur le marché avec des partenaires d’imagerie et dirige le développement de solutions pour accélérer la recherche en imagerie médicale basée sur l’IA/ML dans le cloud. Il est passionné par l’intégration des frameworks ML open source avec la pile AWS AI/ML.
